전자폐수의 탈질에 있어 다양한 외부탄소원 성능비교 및 미생물 군집변화
Performance and Microbial Community analysis on denitrification of electronic wastewater using various External Carbon Source
Article information
Abstract
목적
전자폐수의 생물학적 탈질과정에서 다양한 종류의 외부탄소원을 적용한 성능비교와 NGS분석을 통해 탈질 전ᆞ후 박테리아 군집변화를 해석하였다.
방법
6종류의 외부탄소원을 선정 후 C/N비 4의 무산소 조건에서 6시간 동안 질산성 질소의 농도를 분석하며 탈질속도를 평가하고 NGS분석을 통해 탈질 전ᆞ후 박테리아의 군집분포변화를 확인하였다.
결과 및 토의
다양한 외부탄소원의 탈질성능 비교 결과 Ethylene glycol(EG)이 6시간 이후 79.9%로 가장 우수하였으며 비탈질속도(SDNR)는 1.000mg NO3--N removal/g MLVSS·hr이었다. NGS를 이용한 박테리아 군집변화는 문(Phylum)수준에서 10개 문이 90% 이상 분포하며 Proteobacteria, Saccharibacteria, Chloroflexi의 순으로 우점 하였고 이 중 Saccharibacteria, Chloroflexi가 탈질에 기여하는 박테리아임을 선행연구 고찰을 통해 간접적으로 확인할 수 있었다. 강(Class) 수준에서의 군집분포 해석 결과 투입된 외부탄소원 성상에 따라 실험군간 군집의 차이를 보였으며 γ-proteobacteria의 경우 탄소원을 투입한 실험군에서 모두 증가되었다. 속(Genus) 수준에서 확인된 탈질박테리아 군집비율은 1.27% 미만으로 다양한 박테리아가 탈질에 관여하다는 것을 알 수 있었다.
결론
Ethylene glycol(EG)의 경우 대체 외부탄소원으로 활용 가능 할 것으로 판단되며 Saccharibacteria, Chloroflexi와 같은 다양한 박테리아가 탈질에 기여하며 질소오염원 제거에 관여하는 것을 간접적으로 확인하였다.
Trans Abstract
Objectives
In the biological denitrification process of electronic wastewater, the denitrification performance of various types of external carbon sources and the changes in the bacterial community before and after denitrification were evaluated through NGS analysis.
Methods
After selecting 6 types of external carbon sources, the concentration of nitrate nitrogen was analyzed for 6 hours under anoxic conditions with a C/N ratio of 4 to evaluate the denitrification rate, and the changes in the community distribution of bacteria before and after denitrification were confirmed through NGS analysis.
Results and Discussion
As a result of comparing the denitrification performance of various external carbon sources, Ethylene glycol(EG) was the best at 79.9% after 6 hours, and the specific denitrification rate(SDNR) was 1.000mg NO3--N removal/g MLVSS·hr. The bacterial community change using NGS was distributed more than 90% of 10 phylums at the phylum level, and Proteobacteria, Saccharibacteria, and Chloroflexi were dominant, and among them, Saccharibacteria and Chloroflexi were confirmed to be bacteria contributing to denitrification. At class and genus level, when a external carbon source was added, the number of γ-proteobacteria increased in all experimental conditions, but the distribution of denitrifying bacteria was less than 1.27%, indicating that various bacteria contributed to denitrification.
Conclusion
In the case of Ethylene glycol(EG), it is judged that it can be used as an external carbon source, and there was no significant change in the community depending on the type of carbon source injected, and various bacteria such as Saccharibacteria and Chloroflexi contributed to denitrification and eliminated nitrogen pollutants.
1. 서 론
전자폐수에 포함된 질소오염원의 생물학적 처리과정에서 암모니아(NH3)를 질산이온(NO3-)으로 전환시키는 것은 매우 중요하나 이 과정은 실제로 폐수 내에 어떠한 질소도 제거하지 않는다. 하지만 탈질과정은 질산이온의 환원을 촉진시켜 궁극적으로 질소기체로 최종 처리되는 단계로 산소가 고갈된 무산소(Anoxic)조건에서 일어나는 일종의 호흡과정(Anoxic respiration)인 산화-환원 반응이며 유기물은 전자공여체로 작용하고 질산이온은 산소 대신 전자수용체로 이용하게 된다. 이 과정에 관여하는 탈질 박테리아는 대표적인 4가지 효소인 질산염 환원효소(Nitrate Reductase, Nar), 아질산염 환원효소(Nitrite Reductase, Nir), 산화질소 환원효소(Nitric oxide Reductase, Nor)와 질소 환원 효소(Nitrous oxide Reductase, Nos)를 생성하게 되며 효소의 포함여부에 따라 탈질 미생물로 분류한다(Fig. 1).
탈질 과정에서의 외부탄소원 사용은 박테리아 성장을 촉진하여 효율적인 질소오염원 제거를 가능하도록 하며[2,3] 탈질 속도와 경제성을 고려하여 탄수화물, 알코올, 유기산 염, 아미노산류[4]의 메탄올, 에탄올, 아세테이트, 당밀[5], 아세트산[6] 등이 사용되어지고 있다. 최근에는 다양한 부산물과 폐기물을 재활용하여 보다 환경 친화적이거나 박테리아 적응성과 안전적 측면을 고려하여 처리장 특성에 적합한 외부탄소원을 적용하려는 연구사례가 제시되고 있다[7]. 외부탄소원 투입량은 질산성질소 농도에 상당하는 C/N비(C/N ratio)를 결정하여 공급하며 C/N비가 낮으면 탈질저하로 원활한 질소오염원 제거가 어려우며 C/N비가 높으면 탄소원이 2차오염물질로의 잔류와 운영비가 증가되어 적정한 C/N비를 유지하여야 하며 보통 1.5~5 범위에서 운영 중에 있다[8]. 투입되는 외부탄소원 종류에 따라서도 C/N비는 달라지는데 일반적으로 방향족은 비방향족보다 C/N비가 높아야 하며 비방향족의 경우 완전한 탈질에 필요한 최소 C/N비는 분자량이 증가함에 따라 증가하게 되며[9] 글리세린의 경우 메탄올보다 낮은 C/N비 조건에서 유사한 탈질효율을 보이기도 한다[4].
차세대 염기서열 분석(Next Generation Sequencing, NGS)은 분자생물학 수준에서의 군집분석과 다양성 평가가 가능하기 때문에 생물학적 처리과정에서의 오염원 제거와 슬러지 벌킹처럼 수질변화에 관여하는 박테리아의 규명이 가능하다. 이중에서도 16s rRNA를 기반으로 하는 Amplicon sequencing 방법은 유전자 변이의 가능성이 낮고 Primer로 사용할 수 있는 다수의 보존부위(Conserved regions)를 가지고 있어 PCR (Polymerase Chain Reaction)증폭이 용이하여 박테리아 군집 분석에 적합하며 16S rRNA의 승인된 종(Species)은 1980년 1,791종에서 현재 61,700종 이상까지 증가하여 16s rRNA 기반의 유전자 염기서열 분석연구는 빠르게 향상되었다[11].
본 연구에서는 전자폐수 내 질소오염원을 최종처리하기 위한 탈질과정에서 다양한 종류의 외부탄소원을 적용함에 따라 탈질성능을 비교하며 대체 외부탄소원으로써의 가능성을 확인하고자 하였으며 NGS분석을 통해 탈질 전ᆞ・후 박테리아의 군집변화가 어떻게 변화되는지를 살펴보았다.
2. 실험방법
2.1. 외부탄소원 선정
탈질성능 비교를 위해 선정된 외부탄소원은 총 6종류로 사업장에서의 사용빈도가 높은 Methanol과 상대적으로 독성이 적은 Ethanol, 친환경 측면의 Glucose, 당알코올류의 Ethylene glycol과 Glycerol, Propanol의 이성질체 Isopropyl alcohol를 탄소원으로 선정하였다. Ethylene glycol의 경우 호기성과 혐기성 조건에서 모두 생분해가 가능하며[12] Isopropyl alcohol은 외부탄소원으로의 연구사례는 많이 보고되지 않았으나 1가 알코올(Methanol, Ethanol) 중 3개 탄소를 포함하여 외부탄소원으로 선정하였다.
2.2. 탈질 성능 실험
탈질성능 평가에 이용될 폐수는 A전자회사 질산화조 마지막 반응조내의 내부반송 슬러지(Return Activated Sludge, RAS)를 대상으로 하였으며 본 실험에 앞서 성상분석을 실시하였다. 질산성 질소(NO3--N)의 분석은 C-mac사 분석키트를 410nm 파장에서 분석하였으며 암모니아성 질소(NH3+-N)는 HACH(USA)사 Nessler 시험방법으로 425 nm 파장에서 분석하였다. 또한, 외부 탄소원의 화학적산소요구량(Chemical Oxygen Demand, CODcr)은 HACH(USA)사의 분석키트를 사용하여 620nm 파장에서 분광광도계 DR-6000(HACH, USA)을 이용하여 측정하였다. 각 분석에 사용된 총유기탄소(Total Oxygen Carbon, TOC)와 총질소(Total Nitrogen, T-N) 분석은 TOC-L analyzer series(Shimadzu Co. LTD., Japan)으로 수행하였다. 추가로 MLSS(Mixed Liquor Suspended Solids)와 MLVSS(Mixed Liquor Volatile Suspended Solids) 분석으로 내부반송 슬러지 내의 미생물의 농도를 확인하였다. 탈질성능 평가를 위해 내부반송 슬러지의 초기 질산성질소와 C/N비 4에 상당하는 외부탄소원 CODcr값을 기준으로 투입 후 Jar-test를 이용하여 60 rpm으로 6시간 동안 무산소조건에서 회분식 실험을 진행하며 시간변화에 따른 질산성 질소 농도의 감소를 확인하였다.
투입된 외부 탄소원 종류에 따른 탈질 성능평가는 비탈질율(Specific Denitrification Rate, SDNR)을 이용하여 산출하였으며 SDNR은 식 (1)과 같이 질소제거 반응조에서 단위시간, 미생물량당 제거되는 질산성 질소량을 의미하며 NO0은 초기 질산성질소의 농도(mg/L), NO는 반응종료 후 질산성질소 농도(mg/L), θ는 반응시간(hr), MLVSS는 미생물의 농도(mg/L)를 나타내며 SDNR이 높을수록 탈질 반응속도가 빠르다는 것을 의미한다. 본 실험에 이용된 내부반송 슬러지와 외부탄소원 6종류에 대한 성상은 Table 1, 2와 같다.
2.3. 미생물 군집해석
다양한 외부탄소원 투입에 따라 변화되는 박테리아의 군집을 살펴보기 위해 NGS(Next Generation Sequencing)분석 통해 확인하였으며 탄소원을 투입하지 않은 RAS와 탈질 실험 종료 후 RAS를 대상 박테리아 군집분포변화 살펴보았다.
Exgene™ Stool DNA mini(GeneAll, Korea)를 이용하여 추출한 metagenomic DNA 중 박테리아의 16S ribosome DNA libraries V4 region 증폭을 위하여 515F(GTGCCAGCMGCC GCGGTAA) 및 806R(GGACTACHVGGGTWTCTAAT) 서열과 함께 Illumina overhang adapter 서열이 부착된 primer를 사용하였다[13]. 이어서 각각의 primer와 추출한 5ng/µL DNA, DNA polymerase인 2x KAPA HiFi HotStart ReadyMix(KAPA BIOSYSTEMS, USA)를 첨가하여 1st PCR을 수행하였다. 1st PCR은 95°C에서 pre-denaturation 3분, 95°C에서 denaturation 30초, 55°C에서 primer annealing 30초, 72°C에서 extension 30초를 25회 반복하고, 72°C에서 final extension 5분을 진행하였다. 1st PCR을 통해 생성된 free primer와 primer dimer 중에서 16S V4 amplicon을 정제하기 위해 AMPure XP beads(Beckman Coulter, USA)를 사용하였다. 1st PCR 과정 후 primer에 dual indices와 Illumina sequencing adapters를 부착하기 위해 Nextera XT DNA Library Preparation Kit (Illumina, USA)를 사용하였으며 2x KAPA HiFi HotStart ReadyMix와 함께 첨가하여 2nd PCR을 실시하였다. 2nd PCR 반응조건 95°C에서 predenaturation 3분, 95°C에서 denaturation 30초, 55°C에서 primer annealing 30초, 72°C에서 extension 30초를 8회 반복, 마지막으로 72°C에서 final extension 5분을 수행하여 증폭을 중단하였다. 2nd PCR 후 염기서열 정량을 위해 AMPure XP beads를 사용하여 정제하였다.
PCR을 통해 생성된 염기서열은 Agarose 1% 겔에서 전기영동(Electrophoresis)으로 bp(Base pair) 크기를 확인하며 E-Gel™ 1Kb Plus Express DNA Ladder(Thermos, USA)를 마커 (Maker)로 하여 bp 크기를 비교하였다. 전기영동을 통해 모든 샘플이 일정한 bp를 나타나는 것을 확인 후 Qubit 장비를 이용하여 염기서열의 농도를 확인하며 각 샘플 간의 농도를 동일하게 희석하여 하나의 샘플로 혼합 후 iSeq100 Reagent (Illumina, USA)에 주입하여 illumina iSeq 100 (Illumina, USA) 장비를 통해 염기서열의 분석과 EzbioCloud(CJ Bioscience, Korea)의 16S-based MTP를 통해 박테리아 군집을 해석하였다.
3. 결과 및 고찰
3.1. 외부탄소원 별 탈질성능 특성
다양한 외부탄소원을 투입 후 6시간 동안 탈질성능을 비교한 결과 Ethylene glycol(EG)을 사용한 실험조건에서 79.9%로 가장 우수하였으며 Glycerol(G), Ethanol(E), Isopropyl alcohol (IPA), Glucose(GC), Methanol(M)의 순서로 73.2%, 62.0%, 61.4%, 43.6%, 41.7%의 탈질성능을 보였다(Fig. 2, Left). 6시간 반응 후의 비탈질속도(SDNR)를 산출한 결과에서는 EG를 첨가한 실험군에서 1.000mg NO3--N removal/g MLVSS·hr로 가장 높은 동역학적 인자를 도출하였으며 외부탄소원 종류에 따라 0.512~1.000mg NO3--N removal/g MLVSS·hr 범위의 SDNR를 갖는 것으로 나타났다(Fig. 2, Right). Grath M. Horne [14]의 연구결과로 제시한 SDNR의 경우 0.332~3.328mg NO3--N removal/g MLVSS·hr의 범위로 본 연구와 동일한 Bardenpho 공법과 비교해 볼 때 유사한 범위의 SDNR을 보였으나 외부탄소원의 종류에 따라 다양한 범위의 SDNR이 도출되는 것 또한 알 수 있었다.

Denitrification efficiency of using various external carbon source(Left), Comparison of SDNR input external carbon source after 6hrs(Right).
높은 C/N비에 따른 과잉의 외부탄소원 투입은 2차 오염원으로 작용함에 따라[15,16] 실험 종료 후 CODcr의 잔류농도를 확인한 결과 M에서 83.5mg/L로 가장 많은 탄소원이 잔류하였으며 EG, IPA, GC, E, G을 투입한 실험조건에서는 33.9~ 38.4 mg/L 농도로 분포하는 것을 알 수 있었다(Table 3). 실험초기 C/N비 4에 해당하는 외부 탄소원 투입농도 93.6 mg/L와 내부반송 슬러지에 포함된 CODcr의 농도를 고려해볼 때 외부탄소원의 67.8% 이상을 소비하였으며 탈질효율이 가장 낮은 M에서 29.9%의 외부탄소원의 소비가 덜 일어나 동일한 C/N비 투입조건에서도 37.9%의 차이가 발생하는 것을 확인하였다. 한편, 포도당의 경우 탈질성능에 비해 잔류 CODcr가 낮아 탈질에 관여하는 박테리아 이외의 박테리아로부터 세포합성에 필요한 탄소원으로 이용되었을 것으로 판단된다.
3.2. 박테리아 군집 분석
탈질과정에서 변화되는 박테리아 군집변화를 NGS(Next Generation Sequencing)를 사용하여 살펴보았으며 박테리아 분류(Taxonomy)에 이용된 Read counts는 94,272~95,082개로 유의성(Significance) 있는 수준의 Read counts를 4,000개 이상으로 고려해 볼 때[17] 본 연구연구의 군집분석을 위해 산출된 Read counts는 유의성 있는 수준의 결과로 볼 수 있으며 외부탄소원을 투입한 실험군 모두에서 Reads counts는 8.4~9.1% 증가하여 탈질과정에서의 박테리아 개체수 또한 증가된 것을 알 수 있었다(Table 4).
3.2.1. 문(Phylum) 수준의 박테리아 군집변화
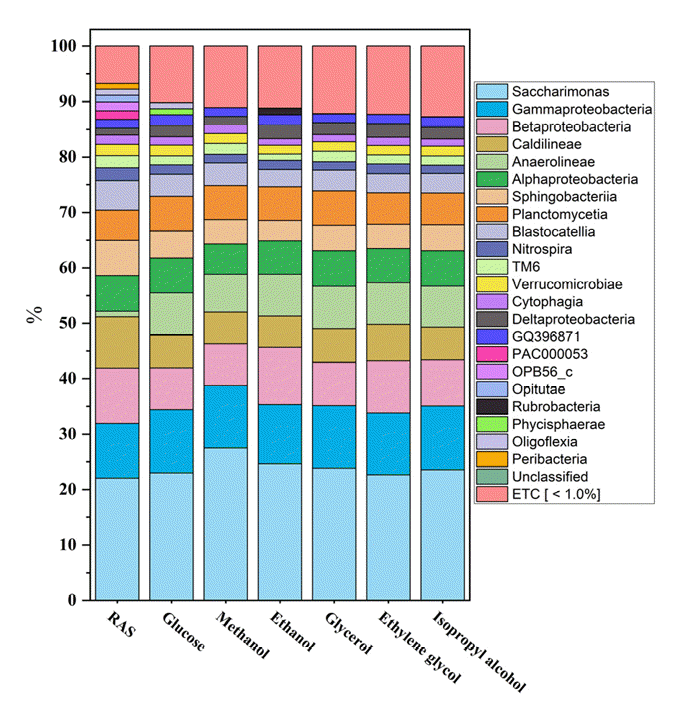
문(Phylum)수준에서 분류(Taxonomy)된 전체 44개 문 박테리아 중 상위 10개 문의 수가 90%이상 분포하였으며 외부탄소원을 투입하지 않은 대조군과 비교해 볼 때 군집의 분포는 크게 변화되지는 않았다. 가장 큰 우점을 차지하는 문은 Proteobacteria로 28.3~30.2%였으며 다음으로는 Saccharibacteria가 22.6~26.7%, Chloroflexi는 12.0~16.2%, Planctomycetes 6.1~7.6%, Bacteroidetes 5.6~8.5% 순으로 군집분포를 이루고 있었다(Fig. 3). 가장 높은 군집분포를 갖는 Proteobacteria는 폐수 내 다양한 환경조건에서 출현되는 박테리아이며[18] Saccharibacteria의 경우 호기성 및 무산소조건 모두에서 탈질에 영향을 주는 박테리아임을 시사하는 연구 결과가 보고되고 있다[19]. 또한, 외부 탄소원 투입 후 가장 큰 군집 변화가 일어난 문인 Chloroflexi는 유전자 및 효소 구조분석(Gene and enzyme structural analysis)을 통해 Nitrite reductase(Nir)를 확인함으로써 탈질에 관여함이 밝혀져 있어 본 실험에서도 Chloroflexi는 탈질에 관여한 것으로 추정하고 있다[20]. 따라서 Proteobacteria, Saccharibacteria, Chloroflexi가 차지하는 군집비율은 생물학적 탈질과정에서의 일반적 특징을 보이는 것으로 해석된다.
3.2.2. 강(Class) 수준의 박테리아 군집변화
각 실험군에 대한 NGS 분석결과를 좀 더 세부적으로 관찰하기 위해 강(Class)수준에서 분류된 102 종류의 박테리아를 대상으로 군집변화 결과를 Fig. 4에 나타내었다. 탄소원이 투입된 실험군 모두에서 0.6~5.5%까지 군집이 증가하였으며 Saccharibacteria 속의 Saccharimonas가 22.7~27.5%로 가장 큰 분포로 우점 하였다. 다음으로는 문(phylum)에 속한 Proteobacteria 중 강(Class) 수준에서 알파(α), 베타(β), 감마(γ) Proteobacteria를 구분하여 살펴본 결과 α-proteobacteria가 5.4~6.3%로 외부 탄소원을 투입한 실험군에서 군집비율이 감소하였으며 β-proteobacteria 역시 GC를 제외한 나머지 실험군에서 감소하는 것을 확인할 수 있었다. 그러나 γ-proteobacteria의 경우 외부탄소원을 투입한 실험군 모두에서 군집비율이 증가하여 본 실험의 탈질 조건에서는 Proteobacteria 중에서도 감마(γ)집단의 박테리아군이 우점하는 것으로 나타났다. 베타(β)집단에 속한 박테리아들의 경우 비교적 안정적인 운전조건의 슬러지내에 우점하며 감마(γ)집단의 박테리아는 일반적인 배지로 배양이 가능한 세균들로 알려져 있다[21,22].
3.2.3. 탈질 박테리아 세균군집구조
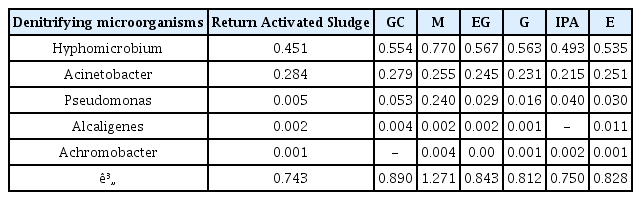
탈질박테리아(Denitrifying bacteria)는 질산염(NO3-)을 질소가스(N2)로 환원시키며 Hyphomicrobium, Achromobacter, Acinetobacter, Alcaligenes, Chromobacterium, Pseudomonas, Moraxella, Micrococcus, Corynebacterium, Propionibacterium 등이 탈질에 관여하는 것으로 알려져 있다[23~29]. 본 연구에서는 탈질박테리아와 탈질 효율과의 상관성을 확인하기 위해 군집변화를 속(Genus) 수준에서 살펴보았다(Table 6). 그 결과, 속(Genus) 수준에서 분류된 박테리아 총 735개 속 중 탈질박테리아는 Achromobacter, Acinetobacter, Alcaligenes, Hyphomicrobium, Pseudomonas 총 5개 속이었으며 외부탄소원 투입 전 탈질박테리아 분포는 전체 군집분포의 0.74%이었으나 외부탄소원을 투입 후 탈질 박테리아의 군집비율은 다소 증가되는 것을 알 수 있었다. 특히 M을 탄소원으로 사용하였을 때 Hyphomicrobium 속의 군집비율이 크게 증가한 것은 보통 탈질계에서 주요 역할을 한다는 다른 연구결과와 유사하며[29] Hyphomicrobium 속 박테리아는 Methylotrophs로 추정된다. 또한, Pseudomonas의 군집비율이 증가한 것은 Psedomonas aeruginosa에서 게놈(Genome)분석을 통해 Methanol의 이용과 완전한 탈질대사에 필수적인 유전자 세트의 존재를 확인함으로써 탈질 Methylotrophic 박테리아임을 밝혀 낸 점을 고려해 볼 때 Peudomonas 속이 탈질에 관여하는 것으로 보인다[30]. 그러나 탈질박테리아가 차지하는 군집비율은 매우 낮았으며 탈질 효율이 우수 할 수록 탈질박테리아의 분포가 비례하여 증가되는 경향 또한 확인할 수 없었다. 이러한 결과는 현재까지 규명된 탈질박테리아 외에도 다양한 박테리아들이 다른 대사경로를 통해 질소오염원을 제거하는데 관여한다는 것을 시사한다[31].
3.3. 박테리아 군집 다양성 해석
각 실험군으로부터 도출된 군집분포의 다양성(Diversity)과 풍부도(Richness)를 Shannon과 Chao1 지수를 이용하여 확인하였으며 풍부도의 경우 1,642~1,698의 범위로 탄소원을 투입한 모든 실험군에서 증가되었으며 GC를 투입한 실험조건에서 6.2%까지 가장 크게 증가하였다. Shannon 지수 또한 외부탄소원이 투입된 모든 실험군에서 4.97~5.26의 범위로 최대 5.5%까지 증가하였으며 GC를 투입한 실험조건에서 가장 큰 다양성의 증가를 보였으나 종합적으로 고찰해 볼 때 풍부도가 증가할수록 다양성 또한 비례하여 증가하는 상관성은 나타나지 않았다(Table 7).
박테리아의 군집간 유사도(Similarity)해석을 위해 주성분 분석(Principal Coordinates Analysis, PCoA)방법을 이용하여 강(Class)수준에서 분석한 결과 외부탄소원으로 M, IPA, E, EG를 사용한 실험군이 근접하게 분포되며 높은 유사도를 보였으며 GC와 G는 외부탄소원을 투입하지 않은 실험군에서는 군집간 거리가 멀어 유사도가 비교적 낮은 것으로 나타나 실험군 간 군집의 차이를 확인할 수 있었다(Fig. 5). A군집 특성을 살펴보면 OH-의 수가 1개 또는 2개로 이루어진 알코올 급수가 1가 또는 2가로 이루어진 형태의 외부탄소원이며 G는 OH-의 수가 3개인 Trihydric 알코올 인 것을 알 수 있다. 또한, GC의 경우 피라노스형태의 고리구조로 이루어진 것을 고려해 볼 때 투입된 외부탄소원의 화학적 구조에 따라 군집간 유사도의 변화가 일어나는 것을 추론할 수 있었다.
4. 결론
본 연구는 전자폐수 내 포함된 질소오염원을 처리하는 최종 단계인 생물학적 탈질과정에서 이용되는 다양한 종류의 외부탄소원을 투입하며 탈질성능 비교와 NGS분석을 통해 박테리아의 군집분포 변화와의 상관성을 살펴보았다.
1. 외부탄소원 투입 후 6시간까지의 탈질실험 결과, EG에서 탈질효율은 79.9%, 비탈질속도는 1.000 NO3--N mg removal/g MLVSS·hr로 가장 우수하여 6종류의 실험군 중 외부탄소원으로의 사용 가능성이 가장 높았다.
2. NGS분석을 통해 박테리아 군집변화를 문(Phylum), 강(Class)수준에서 살펴보았으며 전체 44개 문 중 상위 10개의 문이 90%를 차지하며 이중에서 26.7%와 16.2%를 차지하는Saccharibacteria와 Chloroflexi 문은 탈질에 기여하는 박테리아임을 간접적으로 알 수 있었으나 탄소원을 미투입한 대조군과 비교해 볼 때 기대할 만한 수준의 군집변화는 일어나지 않았다.
3. 속(Genus)수준에서 확인된 탈질박테리아는 5개 종으로 M을 투입한 실험조건에서 1.27%로 가장 큰 군집변화가 일어났으나 탈질효율과는 반비례하는 결과를 나타내어 다양한 박테리아가 탈질에 관여한다는 것을 추론할 수 있었으며 가장 큰 우점을 차지하는 속은 Hyphomicrobium으로 대조군(0.451%)과 비교했을 때 EG(0.567%)에서 군집비율은 다소 증가되었지만 Hyphomicrobium sp. strain이 EG에서 증식에 실패했다는 결과와는 차이를 보이는 결과라 할 수 있다[32]. 그러나 포도당을 사용한 결과와 비교해 볼 때 군집비율이 상위에 있는 Hypermicrobium 및 Acinetobacter 군집의 증가는 적어도 EG가 포도당을 대체할 만한 탄소원으로의 가능성은 충분하다고 볼 수 있다.
4. 탈질 실험군 간 박테리아 유사도는 강(Class) 수준에서 차이를 나타내었으며 투입된 외부 탄소원의 알코올 급수를 결정하는 OH-의 수가 군집간 유사도에 영향이 있는 것으로 판단된다.
Notes
Declaration of Competing Interest
The authors declare that they have no known competing interests or personal relationships that could have appeared to influence the work reported in this paper.